Appearance
物理信息神经网络
常微分方程求解
考虑如下 Lotka-Volterra system
取参数
步骤 1 导库
导入必要的库,设置好问题类型
python
import torch
import nets
mdl = nets.PINN([1, 20, 10, 2])
mdl.set_obj("ode")我们可以通过上面的语句方便的构建一个 PINN,其中 [1,20,10,2] 的含义是:
- 1:输入神经元的数量为 1,对应问题的自变量
- 20: 第一个隐藏层的神经元数量为 20
- 10:第二个隐藏层的神经元数量为 10
- 2:输出神经元的数量为 2,对应问题的
语句mdl.set_obj("ode")指定求解问题为常微分方程(组)
步骤 2 翻译
将求解问题翻译成代码。我们用符号 u 来代表神经网络的输出,du 来代表神经网络输出对输入的导数,那么,
- u[0] 就代表本例的
- u[1] 就代表本例的
- du[0] 代表
- du[0] 代表
于是我们的问题被翻译为
python
t0 = torch.tensor([[0.0]])
u0 = torch.tensor([[1.2,1.]])
def conditions(t, u, du):
f1 = 0.5*u[0]-0.5*u[0]*u[1] - du[0]
f2 = -2*u[1] + 2*u[0]*u[1] - du[1]
f3 = u0 - mdl(t0)
return f1, f2, f3我们需要在函数 conditions 返回各条件的残差,f1, f2 对应方程的两个条件,f3 对应初值条件。conditions 的三个参数依次代表神经网络输入、神经网络输出和神经网络输出对输入的导数。
步骤 3 传条件
将翻译好的条件传给 PINN。
python
mdl.conditions = conditions这里 mdl.conditions 的名字不能更改,但右端的 conditions 是可以修改的。比如我们定义 residual,用 x 代表网络输出,y 代表网络输出,dy 代表网络输出对输入的导数,也就是我们定义了如下函数:
python
t0 = torch.tensor([[0.0]])
u0 = torch.tensor([[1.2,1.]])
def residual(x, y, dy):
f1 = 0.5*y[0]-0.5*y[0]*y[1] - dy[0]
f2 = -2*y[1] + 2*y[0]*y[1] - dy[1]
f3 = u0 - mdl(t0)
return f1, f2, f3那么我们可以将 residual 传给 PINN,效果将完全相同:
python
mdl.conditions = residual步骤 4 训练
选择配置,进行训练。我们的训练函数是 pde_solve,当我们调出这个函数时,编辑器会自动弹出一份文档,里面有可参考的配置信息,我们也可以直接复制:
python
x = torch.linspace(0, 6, 100, requires_grad=True).view(-1, 1)
sets = {
"x": x,
"optimizer": torch.optim.LBFGS,
"lr": 0.1,
"epochs": 50,
}
mdl.pde_solve(**sets)这里的 x 是用于训练的数据点,对应我们本例问题自变量
步骤 5 可视化
可视化。上面的训练在 i5 12 代的笔记本上大约需要耗费 2.5s. 训练结束后,我们可以使用 predict 函数对求解区间的任意点进行预测,一个简单的可视化代码如下:
python
import matplotlib.pyplot as plt
t = torch.linspace(0, 6, 200).view(-1, 1)
plt.plot(t, mdl.predict(t))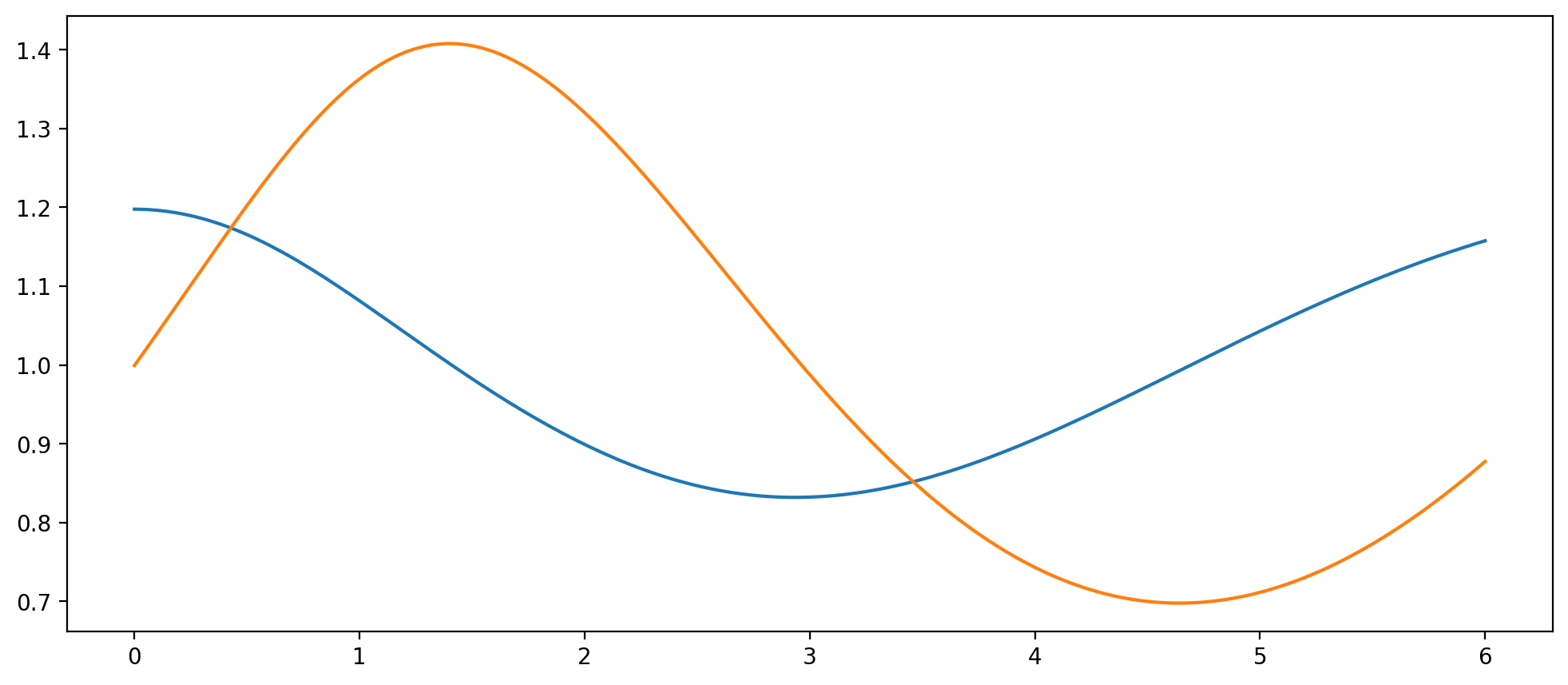
偏微分方程求解
考虑如下 poisson equation:
其真实解为
步骤 1 导库
导入必要的库,设置好问题类型
python
import torch
import nets
import myfuntools as mft
mdl = nets.PINN([2, 20, 10, 1])
mdl.set_obj("pde")我们可以通过上面的语句方便的构建一个 PINN,其中 [2,20,10,1] 的含义是:
- 2:输入神经元的数量为 2,对应问题的自变量
- 20: 第一个隐藏层的神经元数量为 20
- 10:第二个隐藏层的神经元数量为 10
- 1:输出神经元的数量为 1,对应问题的
语句mdl.set_obj("pde")指定求解问题为偏微分方程,由于 PINN 的默认设置就是求解偏微分方程,所以该语句也可以省略不写。
步骤 2 翻译
将求解问题翻译成代码。由于问题有一个方程条件和"四个"边界条件, 所以我们一共需要构建 5 个残差。先从构造采样点开始:
python
be = mft.creat_ereadata(0,1,0,1,500)
b0 = mft.creat_ereadata(0,0,0,1,50)
b1 = mft.creat_ereadata(0,1,0,0,50)
b2 = mft.creat_ereadata(1,1,0,1,50)
b3 = mft.creat_ereadata(0,1,1,1,50)这样我们就得到了区域
python
f0 = b0[:, 1:2] ** 3
f1 = b1[:, 0:1] * torch.exp(-b1[:, 0:1])
f2 = (b2[:, 1:2] ** 3 + 1) / torch.e
f3 = (b3[:, 0:1] + 1) * torch.exp(-b3[:, 0:1])
fe = torch.exp(-be[:, 0:1]) * (be[:, [0]] - 2 + be[:, [1]] ** 3 + 6 * be[:, [1]])从而这四个边界处的残差为:
python
r0 = mdl(b0) - f0
r1 = mdl(b1) - f1
r2 = mdl(b2) - f2
r3 = mdl(b3) - f3现在还差一个区域内的残差,在区域内,我们需要对神经网络输出进行拉普拉斯运算,这设计输出的二阶偏导数,使用下面函数可以方便的构建二阶偏导:
python
mdl.set_diff_chain("u_11", "u_22")其中 "u_" 是固定前缀,"11" 代表对第一个自变量求导 2 次,即
python
mdl.set_diff_chain("u_122333")上面的导数仅仅是一个引用,我们可以在自定义的 conditions 函数中通过 du["u_..."] 来使用我们的导数(du 的名字可以更改):
python
def conditions(x, u, du):
re = du["u_11"] + du["u_22"] - fe
r0 = mdl(b0) - f0
r1 = mdl(b1) - f1
r2 = mdl(b2) - f2
r3 = mdl(b3) - f3
return re, r0, r1, r2, r3步骤 3 传条件
将翻译好的条件传给 PINN。
python
mdl.conditions = conditions步骤 4 训练
选择配置,进行训练。我们的训练函数是 pde_solve,当我们调出这个函数时,编辑器会自动弹出一份文档,里面有可参考的配置信息,我们也可以直接复制:
python
sets = {
"x": be.requires_grad_(True),
"optimizer": torch.optim.LBFGS,
"lr": 1,
"epochs": 100,
}
mdl.pde_solve(**sets)这里的 x 是用于训练的数据点,对应我们本例问题自变量
步骤 5 可视化
可视化。上面的训练在 i5 12 代的笔记本上大约需要耗费 2.5s. 训练结束后,我们可以按需对其可视化:
python
import matplotlib.pyplot as plt
from matplotlib.colors import Normalize
def myplot(mdl):
norm = Normalize(vmin=0, vmax=1)
t1 = torch.linspace(0, 1, 90).view(1, -1)
t2 = torch.linspace(0, 1, 100).view(-1, 1)
acc = torch.exp(-t1) * (t1 + t2**3)
shape = (t1 + t2).shape
p1 = t1.broadcast_to(shape)
p2 = t2.broadcast_to(shape)
pe = torch.cat((p1.flatten().view(-1, 1), p2.flatten().view(-1, 1)), dim=1)
pre = mdl.predict(pe)
fig = plt.figure(figsize=(12, 3))
ax1 = fig.add_subplot(1, 3, 1)
im1 = ax1.imshow(acc, extent=[0, 1, 0, 1], origin="lower", norm=norm)
plt.colorbar(im1)
ax2 = fig.add_subplot(1, 3, 2)
im2 = ax2.imshow(pre.view_as(acc), extent=[0, 1, 0, 1], origin="lower", norm=norm)
plt.colorbar(im2)
ax3 = fig.add_subplot(1, 3, 3)
im3 = ax3.imshow(abs(pre.view_as(acc) - acc), extent=[0, 1, 0, 1], origin="lower")
plt.colorbar(im3)
plt.tight_layout()
myplot(mdl)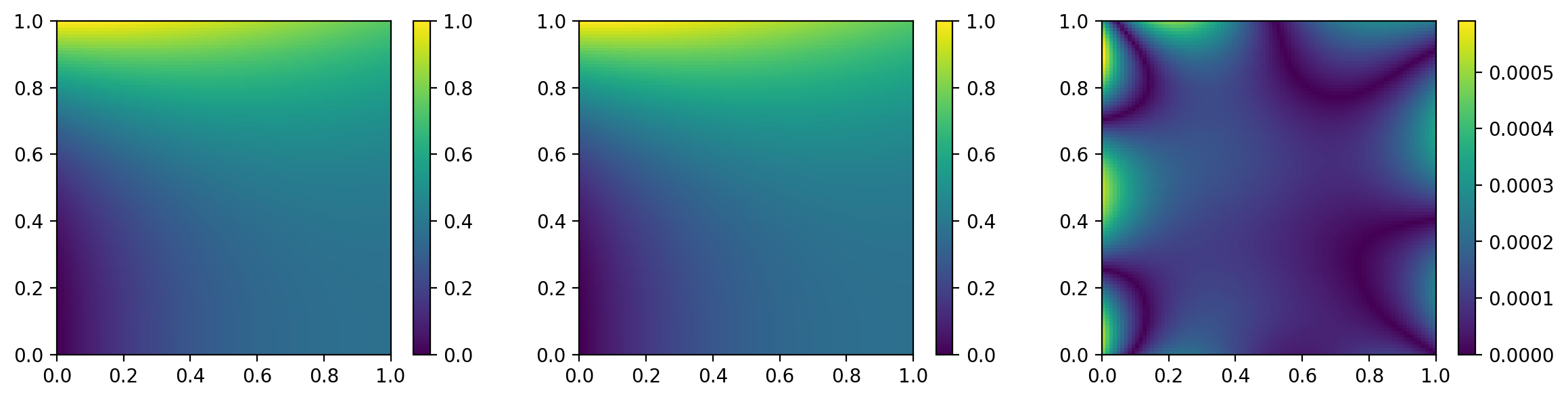 上面从左到右依次是真实值、预测值、逐点绝对误差。
上面从左到右依次是真实值、预测值、逐点绝对误差。